主要职责
中国科学院贯彻落实党中央关于科技创新的方针政策和决策部署,在履行职责过程中坚持党中央对科技工作的集中统一领导。主要职责是:
一、开展使命导向的自然科学领域基础研究,承担国家重大基础研究、应用基础研究、前沿交叉共性技术研究和引领性颠覆性技术研究任务,打造原始创新策源地。 更多+
院况简介
中国科学院是国家科学技术界最高学术机构、国家科学技术思想库,自然科学基础研究与高技术综合研究的国家战略科技力量。
1949年,伴随着新中国的诞生,中国科学院成立。建院70余年来,中国科学院时刻牢记使命,与科学共进,与祖国同行,以国家富强、人民幸福为己任,人才辈出,硕果累累,为我国科技进步、经济社会发展和国家安全作出了不可替代的重要贡献。 更多+
院领导集体
科技奖励
科技期刊
科技专项
科研进展/ 更多
工作动态/ 更多
工作动态/ 更多
中国科学院学部
中国科学院院部

语音播报

反刍动物(如牛、羊、鹿等)依赖其复杂的胃肠道微生物群落消化纤维类物质,合成挥发性脂肪酸和微生物蛋白,同时排放温室气体甲烷。
近日,中国科学院亚热带农业生态研究所整合了来自7种反刍动物10个胃肠道区域的373个宏基因组样本,结合二代与三代测序技术,成功构建了首个反刍动物胃肠道病毒组目录(RGVC)。该目录包含近4.4万个病毒分类单元。团队成功鉴定出大量由长读长技术捕获的超大基因组病毒,极大地拓展了人们对反刍动物肠道病毒世界的认知边界。
研究显示,病毒群落组成并非随机分布,而是呈现出鲜明的空间异质性,这种差异主要取决于胃肠道所在的生理区段,而不是反刍动物宿主种类。进一步通过生物信息学分析,团队发现超过4600个原核生物宿主与近6000个病毒之间上万对的高置信度匹配,揭示了病毒与宿主之间广泛而复杂的互作网络。
深入分析表明,胃肠道病毒的丰度与其对应宿主的丰度呈现高度一致性,不同胃肠道之间的原核微生物群落差异是导致病毒呈现空间异质性的主要原因。近半数的病毒倾向于采用溶原性生活方式,即将其基因组整合到宿主染色体中与之长期共存,且这种生活方式的比例在不同胃肠道区域中呈现规律性变化。
病毒携带的辅助代谢基因广泛参与碳水化合物降解、能量代谢等关键途径,具有重要的代谢调控潜力,且在不同胃肠段间呈差异分布。团队通过基因邻域分析和蛋白质三维结构模拟,排除了宿主基因污染,验证了这些AMGs的病毒来源及其功能的可靠性。这些结果表明,溶源病毒与宿主建立了互利共生关系,共同调节反刍动物的营养吸收与能量获取。
研究首次从“空间异质性”这一生态学核心视角出发,系统揭示了反刍动物胃肠道病毒群落的分布规律、生存策略与功能贡献,拓展了人们对宿主—微生物—病毒互作的认识,为提升饲料转化效率、定向减少甲烷排放,提供了新的科学路径与潜在调控靶点。
相关研究成果发表在Journal of Advanced Research上。研究工作得到国家自然科学基金和国家重点研发计划等的支持。
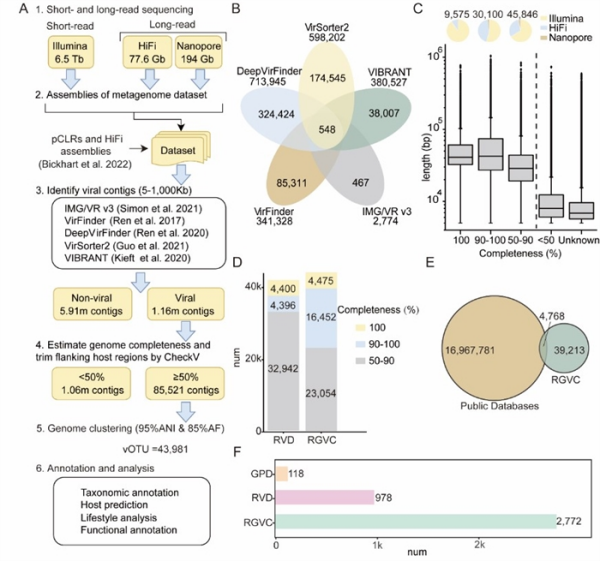
RGVC数据库的构建及特征概述
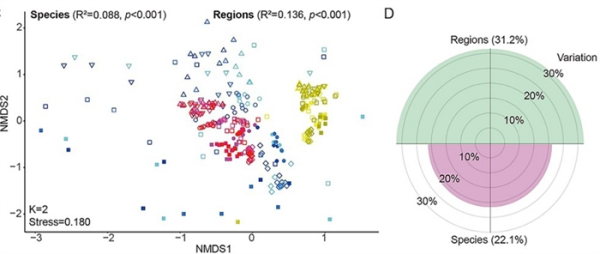
不同胃肠道区域和反刍动物物种的病毒多样性及解释贡献度占比
扫一扫在手机打开当前页

© 1996 - 中国科学院 版权所有 京ICP备05002857号-1  京公网安备110402500047号 网站标识码bm48000002
京公网安备110402500047号 网站标识码bm48000002
地址:北京市西城区三里河路52号 邮编:100864
电话: 86 10 68597114(总机) 86 10 68597289(总值班室)














