主要职责
中国科学院贯彻落实党中央关于科技创新的方针政策和决策部署,在履行职责过程中坚持党中央对科技工作的集中统一领导。主要职责是:
一、开展使命导向的自然科学领域基础研究,承担国家重大基础研究、应用基础研究、前沿交叉共性技术研究和引领性颠覆性技术研究任务,打造原始创新策源地。 更多+
院况简介
中国科学院是国家科学技术界最高学术机构、国家科学技术思想库,自然科学基础研究与高技术综合研究的国家战略科技力量。
1949年,伴随着新中国的诞生,中国科学院成立。建院70余年来,中国科学院时刻牢记使命,与科学共进,与祖国同行,以国家富强、人民幸福为己任,人才辈出,硕果累累,为我国科技进步、经济社会发展和国家安全作出了不可替代的重要贡献。 更多+
院领导集体
科技奖励
科技期刊
科技专项
科研进展/ 更多
工作动态/ 更多
工作动态/ 更多
中国科学院学部
中国科学院院部

语音播报

生物传感器是在各种应用中快速检测、实时成像和精确定量目标分子的重要工具。传统的基因编码生物传感器的底物结合域通常局限于非催化蛋白质如转录因子、周质结合蛋白和G蛋白偶联受体等,而在底物/效应物结合时经历显著构象变化的酶类蛋白质很少被考虑用于基因编码生物传感器的开发。目前,特定有机酸和氨基酸的精确测定通常需要借助高效液相色谱、气相色谱以及质谱等精密仪器,并需要在专业实验室中进行操作,这限制了其在实际应用中的通用性。尤其是在需要即时检测(point-of-care testing,POCT)的情景下,传统的检测方法显得不够实用。因此,亟需开发一种系统性的方法来设计和生成可基因编码的生物传感器,以实现对有机酸及其衍生物的快速、精确检测,并将其应用于各种实际场景。
近日,中国科学院天津工业生物技术研究所高通量编辑与筛选平台实验室研究员王猛带领的科研团队,提出了全新的设计理念并开发了相应的高通量筛选方法,将酶转化为可基因编码的、基于循环排列荧光蛋白(cpFP)的生物传感器(Genetically Encoded Circularly Permuted Fluorescent Protein-based Indicators to Detect Organic Acids,GECFINDER)。
该团队获得了11种有机酸和氨基酸的GECFINDER,并验证了GECFINDERs是针对有机酸和氨基酸的高灵敏度、高特异性生物传感器,具有良好的稳定性和较高的动态范围。该研究进行了GECFINDER应用的初步探索,发掘了基于循环排列荧光蛋白的生物传感器在体外应用的潜力,证明了GECFINDER在复杂生物样品中可以高效、准确地定量氨基酸浓度,有望成为针对苯丙酮尿症(PKU)等先天性代谢疾病的POCT快速诊断工具。此外,该研究还开发了一种基于GECFINDER与液滴微流控相结合的超高通量筛选方法,并筛选了由该团队前期开发的BETTER技术生成的核糖体结合位点突变文库,获得了苯丙氨酸高产菌株(苯丙氨酸产量相较于初始菌株提升了将近10倍)。
该研究将具有催化功能的蛋白应用于基于cpFP生物传感器的开发,构建了将酶快速转化为生物传感器的高通量筛选方法,并探索了基于cpFP生物传感器体外应用的方式,将基于cpFP的生物传感器应用于精确氨基酸定量和与液滴微流控相结合的有机酸高产菌株筛选中,这为将酶作为蓝图用作生物传感器的设计、构建和应用提供了新方向。
相关研究成果发表在《自然-通讯》(Nature Communications)上。研究工作得到国家重点研发计划、国家自然科学基金、合成生物学海河实验室重大攻关类项目、中国科学院青年创新促进会和天津市合成生物技术创新能力提升行动专项的支持。
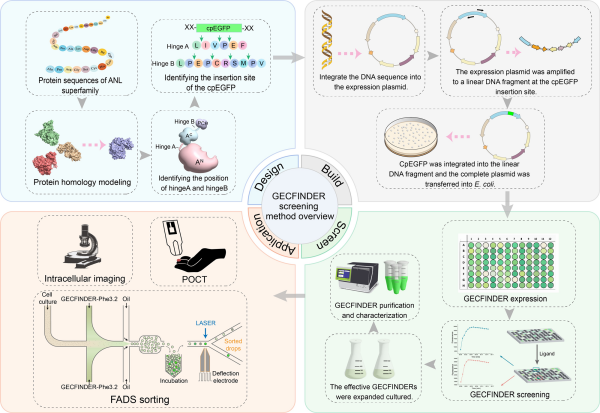
GECFINDER高通量筛选流程示意图

© 1996 - 中国科学院 版权所有 京ICP备05002857号-1  京公网安备110402500047号 网站标识码bm48000002
京公网安备110402500047号 网站标识码bm48000002
地址:北京市西城区三里河路52号 邮编:100864
电话: 86 10 68597114(总机) 86 10 68597289(总值班室)














