
语音播报

9月28日,中国科学院分子植物科学卓越创新中心合成生物学重点实验室研究员张余课题组在Nature Chemical Biology上,在线发表题为CueR activates transcription through a DNA distortion mechanism的研究论文,主要研究细菌Class III转录因子CueR转录激活的分子机制。
大约50年前,法国科学家Jacob和Monod发现乳糖操纵子,首次提出基因表达受到蛋白调控。阻遏蛋白Lac I和代谢物激活蛋白CRP(cAMP receptor protein;也称catabolite activator protein,CAP)被证明能够直接结合乳糖操纵子,分别发挥转录抑制和转录激活的功能。因此,学界对转录因子如何抑制及激活转录产生研究兴趣。大约30年前,Thomas A. Steitz研究组解析出CAP/CRP与DNA的复合物晶体结构,该结构首次展示转录因子识别DNA的方式。在随后的几十年中,科学家们利用化学交联、DNA足迹、遗传突变等方法尝试了解转录因子调控基因转录的具体机制,发现转录因子在启动子DNA的结合位置直接决定其对下游基因的影响,一般来说,转录因子结合在核心启动子区域(-35区和-10区)上游发挥转录激活功能,在核心启动子区域或基因内部则抑制转录。其中,转录激活按照转录因子结合位点距离核心启动子区域远近分为两类,结合位点位于启动子核心区域上游称为第一类转录激活(Class I),结合位点与启动子核心区域稍有重叠称为第二类转录激活(Class II)。2016年、2017年,Richard H. Ebright和Thomas A. Steitz研究组以CAP为模型,在Science上报道细菌Class I与II转录激活因子与RNA聚合酶及启动子DNA的复合物结构,揭示出经典的转录激活分子机制。总体来说,它们通过DNA结合结构域与启动子DNA相互作用,通过其转录激活结构域与RNA聚合酶相互作用,将RNAP聚合酶富集到其调控的启动子DNA区域激活转录。
在探索CAP转录激活机制的同时,David C. Fritzinger发现一种机制特异的转录因子MerR,其能够结合在耐汞基因簇启动子核心区域,与RNAP的结合位置完全重叠,在一般情况下,抑制下游基因表达;胞内汞离子浓度高时,则激活下游基因表达。该现象与上述Class I和Class II的转录激活调控方式完全相悖,因为MerR的结合位置与RNAP结合位置完全重叠,按照此前的规律其应该只发挥转录抑制功能,且MerR调控的基因启动子DNA的-35区和-10区间隔为19bp,而细菌RNA聚合酶只能识别-35区和-10区间隔为17±1的启动子。科研人员在多种细菌中发现该类转录因子的存在,因此该类蛋白被命名为MerR家族转录因子,能够感受胞内的金属离子、氧化状态及抗生素胁迫。自20世纪90年代,科研人员利用DNA足迹手段,发现MerR处于抑制态和激活态时,其结合的启动子DNA构象可能有较大的构象变化。Thomas V. O’Halloran研究组针对MerR家族蛋白进行晶体结构研究,从2003年到2015年,科研人员分别解析CueR apo protein,CueR-DNA二元复合物及CueR-Ag+-DNA三元复合物的晶体结构,阐明该家族成员在不结合配体时,结合标准的B型双链DNA;结合配体后,能够使B型双链DNA发生约90度的弯折,使其局部区域呈现出A型双链DNA的构象,这为该类转录因子的激活机制增加了神秘面纱。鉴于其转录调控方式的特殊性,科研人员将MerR家族转录激活方式命名为非典型的转录激活或Class III转录激活。
为揭示MerR家族转录因子的转录激活机制,张余课题组以大肠杆菌中感应银离子和亚铜离子的CueR蛋白为研究对象,解析CueR、Ag+、启动子DNA及RNA聚合酶的转录激活复合物电镜结构。结果显示,CueR结合在启动子DNA的两个关键区域-35区和-10区之间,使双链DNA在四个位置发生较大程度弯折,特别是位于CueR二聚体中心的位置,DNA发生约90度的弯曲。这种由CueR结合导致的启动子DNA弯曲,使19bp的-35/-10间隔区域重新压缩到17bp的物理距离,从而使RNA聚合酶能够启动下游基因转录。此外,该复合物结构显示,虽然CueR在启动子DNA上的结合位点与RNAP聚合酶的结合位点完全重叠,但是CueR结合在启动子DNA的一侧,而RNAP结合在启动子的另一侧,CueR与RNA聚合酶没有相互作用,二者互不干扰,这一点与Class I及Class II的转录激活机制完全不同。该研究解析以CueR为代表的细菌Class III转录激活复合物结构,揭示该类转录激活蛋白不依赖与RNA聚合酶的相互作用,仅通过改变DNA构象激活转录的分子机制。
张余课题组博士生方城力和美国西北大学博士Steven J. Philips为论文的第一作者。浙江大学医学院研究员冯钰、西北大学教授Thomas V. O’Halloran、张余为论文的通讯作者。研究工作得到浙江大学电镜中心以及国家蛋白质中心(上海)的支持,受到国家自然科学基金、中科院战略性先导科技专项计划(B类)和上海市科技创新行动计划的资助。
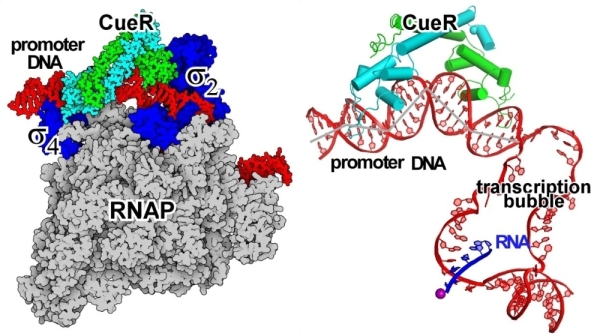
细菌Class III转录激活机制

© 1996 - 中国科学院 版权所有 京ICP备05002857号-1  京公网安备110402500047号 网站标识码bm48000002
京公网安备110402500047号 网站标识码bm48000002
地址:北京市西城区三里河路52号 邮编:100864
电话: 86 10 68597114(总机) 86 10 68597289(总值班室)
